Nuestro grupo se interesa en conocer los mecanismos genéticos y moleculares de la vid, responsables de la regulación de diferentes caracteres relacionados con la calidad de las uvas y el vino. Combinamos la utilización de herramientas vegetales (variedades, clones y cultivos in vitro de vid) y técnicas genéticas y genómicas (secuenciación NGS, RNA-Seq, DNA-seq, qRT-PCR, transformación genética, edición génica CRISPR-Cas), para estudiar fenómenos como la variación genética y fenotípica natural.
Nos hemos focalizado en estudiar el genoma de cultivar ‘Malbec’, que hemos descifrado en el año 2024 (https://doi.org/10.1093/hr/uhae080 http://ibam.mendoza-conicet.gob.ar/resources/genome-visualizer), permitiendo profundizar en aspectos como la base genética del cuajado de la vid, la biología floral o la adaptación de las plantas a condiciones de cambio climático.
- Obtención del genoma de referencia de la variedad ‘Malbec’
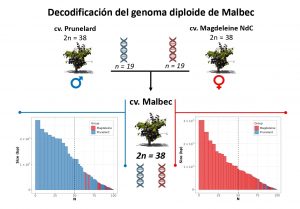 La generación del ensamblado “de novo” del genoma de referencia diploide del cultivar ‘Malbec’ es fundamental para comprender sus atributos varietales y realizar eficientes estudios de edición génica, RNA-seq y DNA-seq. Nuestro equipo de investigación ha descifrado el conjunto del genoma diploide de Malbec, proporcionando una clara perspectiva sobre la interacción genética entre sus dos variedades parentales, Prunelard y Magdeleine Noire des Charentes. Se emplearon técnicas avanzadas de secuenciación de ADN y bioinformáticas para integrar secuencias de ADN largas (PacBio) y cortas (Illumina). De esta manera, se logró un ensamblaje detallado y preciso de los complementos haploides que conforman el genoma diploide de Malbec con tasas de error excepcionalmente bajas. Se develó que más de un cuarto del genoma mostraba regiones polimórficas, lo que lleva a una variación genética significativa. Esta nueva información genómica permitió una profunda evaluación del transcriptoma -la expresión genética activa- de cuatro accesiones clonales de Malbec, cada una con rasgos únicos de composición de las bayas.
La generación del ensamblado “de novo” del genoma de referencia diploide del cultivar ‘Malbec’ es fundamental para comprender sus atributos varietales y realizar eficientes estudios de edición génica, RNA-seq y DNA-seq. Nuestro equipo de investigación ha descifrado el conjunto del genoma diploide de Malbec, proporcionando una clara perspectiva sobre la interacción genética entre sus dos variedades parentales, Prunelard y Magdeleine Noire des Charentes. Se emplearon técnicas avanzadas de secuenciación de ADN y bioinformáticas para integrar secuencias de ADN largas (PacBio) y cortas (Illumina). De esta manera, se logró un ensamblaje detallado y preciso de los complementos haploides que conforman el genoma diploide de Malbec con tasas de error excepcionalmente bajas. Se develó que más de un cuarto del genoma mostraba regiones polimórficas, lo que lleva a una variación genética significativa. Esta nueva información genómica permitió una profunda evaluación del transcriptoma -la expresión genética activa- de cuatro accesiones clonales de Malbec, cada una con rasgos únicos de composición de las bayas.
Los ensamblajes anotados de Malbec están disponibles para visualización y exploración de genes en dos repositorios públicos:
- IBAM-CONICET (http://ibam.mendoza-conicet.gob.ar/resources/genome-visualizer)
- Gramene (https://www.gramene.org)
- Análisis de la variabilidad genética y fenotípica para los componentes del rendimiento en la variedad Malbec
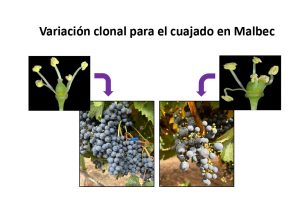 Nos propusimos caracterizar el desempeño reproductivo de 25 selecciones clonales de Malbec cultivadas en condiciones iguales durante diferentes temporadas consecutivas. Evaluamos diferentes rasgos reproductivos en la floración (número de flores por inflorescencia) y en el momento de la cosecha (bayas por racimo). Después de combinar metodologías de fenotipo tradicionales y basadas en imágenes, observamos una amplia diversidad intra-varietal.). Mediante análisis univariados y multivariados agrupamos a los 25 clones en tres grupos divergentes con alta homogeneidad fenotípica dentro de cada grupo, consistente entre las temporadas de cultivo. Este análisis llevó a la identificación de cuatro selecciones clonales con características altamente contrastantes, incluyendo dos clones con características relacionadas con el cuajado de frutas marcadamente comprometidas y dos con mejor rendimiento reproductivo, que se utilizaron como controles. La evaluación en profundidad de estas cuatro selecciones durante la temporada de 2024 nos permitió identificar las posibles causas de su bajo cuajado de frutos, que incluyeron la baja viabilidad del polen y diferentes anomalías de la flor que podrían obstaculizar la polinización de las flores y/o los procesos de fertilización de los óvulos. La identificación y posterior caracterización de estas variantes somáticas de Malbec abren el camino para revelar los mecanismos fisiológicos y moleculares implicados en el cuajado de la vid, una característica relevante para la industria de uva y vino.
Nos propusimos caracterizar el desempeño reproductivo de 25 selecciones clonales de Malbec cultivadas en condiciones iguales durante diferentes temporadas consecutivas. Evaluamos diferentes rasgos reproductivos en la floración (número de flores por inflorescencia) y en el momento de la cosecha (bayas por racimo). Después de combinar metodologías de fenotipo tradicionales y basadas en imágenes, observamos una amplia diversidad intra-varietal.). Mediante análisis univariados y multivariados agrupamos a los 25 clones en tres grupos divergentes con alta homogeneidad fenotípica dentro de cada grupo, consistente entre las temporadas de cultivo. Este análisis llevó a la identificación de cuatro selecciones clonales con características altamente contrastantes, incluyendo dos clones con características relacionadas con el cuajado de frutas marcadamente comprometidas y dos con mejor rendimiento reproductivo, que se utilizaron como controles. La evaluación en profundidad de estas cuatro selecciones durante la temporada de 2024 nos permitió identificar las posibles causas de su bajo cuajado de frutos, que incluyeron la baja viabilidad del polen y diferentes anomalías de la flor que podrían obstaculizar la polinización de las flores y/o los procesos de fertilización de los óvulos. La identificación y posterior caracterización de estas variantes somáticas de Malbec abren el camino para revelar los mecanismos fisiológicos y moleculares implicados en el cuajado de la vid, una característica relevante para la industria de uva y vino.
- Viticultura de Nueva Generación
 El objetivo general de esta línea es estudiar los mecanismos de adaptación de la vid a las condiciones de cambio climático mediante la implementación de una tecnología ‘limpia’ de edición génica CRISPR-Cas9 para vid (Vitis vinifera L), con el propósito de inactivar genes involucrados en la degradación de ABA causada por altas temperaturas. Utilizamos metodologías ‘clásicas’ como la combinación de transformación de embriones somáticos mediada por Agrobacterium tumefaciens o la generación de hairy roots mediada por Rhizobium rhizogenes, con otras más novedosas, como la combinación de la inducción de meristemas de novo mediante reguladores del desarrollo y el uso de nanopartículas para transferir los vectores de transformación.
El objetivo general de esta línea es estudiar los mecanismos de adaptación de la vid a las condiciones de cambio climático mediante la implementación de una tecnología ‘limpia’ de edición génica CRISPR-Cas9 para vid (Vitis vinifera L), con el propósito de inactivar genes involucrados en la degradación de ABA causada por altas temperaturas. Utilizamos metodologías ‘clásicas’ como la combinación de transformación de embriones somáticos mediada por Agrobacterium tumefaciens o la generación de hairy roots mediada por Rhizobium rhizogenes, con otras más novedosas, como la combinación de la inducción de meristemas de novo mediante reguladores del desarrollo y el uso de nanopartículas para transferir los vectores de transformación.
- Impacto fenotípico de una mutación somática floral en el cultivar Listán Prieto
 La accesión Criolla Chica Nº2 (CCN2) es catalogada como una mutación floral del cultivar Criolla Chica (sinónimo de cv. Listán Prieto). Contrariamente a lo que se observa en las variedades hermafroditas cultivadas como Criolla Chica, CCN2 presenta una prevalencia de flores masculinizadas. Con el fin de estudiar la incidencia y las implicaciones fenotípicas de esta mutación, estudiamos plantas CCN2 utilizando Criolla Chica ‘Ballista’ (CCBA) como plantas de control. CCN2 exhibió flores de tipo 1 (masculinizadas) y 2 (intermedias), mientras que el CCBA sólo exhibía flores del tipo 3 (hermafroditas). CCN2 promedia más del 55% de las flores del tipo 1 por inflorescencia y observamos un alto porcentaje de abscisión de inflorescencia por planta (alrededor del 50%), mientras que este fenómeno no se observa en CCBA. Además, las secciones histológicas de flores en diferentes etapas de desarrollo mostraron que para las flores de tipo 1 de CCN2, los tejidos de estilo y estigma muestran desarrollo nulo con óvulos atrofiados; estas estructuras están presentes, aunque mal desarrolladas en las flores del tipo 2, produciendo potencialmente las pocas bayas por racimo observadas en la cosecha. En general, la mutación floral estudiada identificada en CCN2 afecta fuertemente al desarrollo de los tejidos y órganos reproductivos femeninos, obstaculizando drásticamente la tasa de cuajado de frutos y la producción de racimos.
La accesión Criolla Chica Nº2 (CCN2) es catalogada como una mutación floral del cultivar Criolla Chica (sinónimo de cv. Listán Prieto). Contrariamente a lo que se observa en las variedades hermafroditas cultivadas como Criolla Chica, CCN2 presenta una prevalencia de flores masculinizadas. Con el fin de estudiar la incidencia y las implicaciones fenotípicas de esta mutación, estudiamos plantas CCN2 utilizando Criolla Chica ‘Ballista’ (CCBA) como plantas de control. CCN2 exhibió flores de tipo 1 (masculinizadas) y 2 (intermedias), mientras que el CCBA sólo exhibía flores del tipo 3 (hermafroditas). CCN2 promedia más del 55% de las flores del tipo 1 por inflorescencia y observamos un alto porcentaje de abscisión de inflorescencia por planta (alrededor del 50%), mientras que este fenómeno no se observa en CCBA. Además, las secciones histológicas de flores en diferentes etapas de desarrollo mostraron que para las flores de tipo 1 de CCN2, los tejidos de estilo y estigma muestran desarrollo nulo con óvulos atrofiados; estas estructuras están presentes, aunque mal desarrolladas en las flores del tipo 2, produciendo potencialmente las pocas bayas por racimo observadas en la cosecha. En general, la mutación floral estudiada identificada en CCN2 afecta fuertemente al desarrollo de los tejidos y órganos reproductivos femeninos, obstaculizando drásticamente la tasa de cuajado de frutos y la producción de racimos.
- Regulación de la biosíntesis de antocianinas mediada por ARNs largos no codificantes en zanahoria
 Las antocianinas son flavonoides, una clase de compuestos fenólicos sintetizados a través de la ruta de los fenilpropanoides, que confieren pigmentación morada, roja y azul a varios órganos y tejidos de muchas especies de plantas. Estos pigmentos cumplen varias funciones en la planta, incluida la atracción de polinizadores a las flores y los dispersores de semillas a las frutas, la protección contra la radiación ultravioleta, entre otras. La participación de factores de transcripción (TFs) de la familia MYB en la regulación de la biosíntesis de antocianinas en diferentes tejidos vegetales ha sido reportada en diversas especies. Particularmente, los TFs DcMYB7 y DcMYB6 desempeñan un papel clave en el control de la pigmentación de las zanahorias moradas debida a la acumulación de antocianinas. En trabajos previos de nuestro grupo, se identificó dos transcriptos “antisentido” (asDcMYB6 y asDcMYB7) asociados a DcMYB6 y DcMYB7 y pudiendo potencialmente, participar en su regulación. En esta línea de trabajo proponemos probar de manera funcional, el mecanismo por el cual asDcMYB7 estaría ligado a la activación de la biosíntesis de antocianinas mediante el gen DcMYB7.
Las antocianinas son flavonoides, una clase de compuestos fenólicos sintetizados a través de la ruta de los fenilpropanoides, que confieren pigmentación morada, roja y azul a varios órganos y tejidos de muchas especies de plantas. Estos pigmentos cumplen varias funciones en la planta, incluida la atracción de polinizadores a las flores y los dispersores de semillas a las frutas, la protección contra la radiación ultravioleta, entre otras. La participación de factores de transcripción (TFs) de la familia MYB en la regulación de la biosíntesis de antocianinas en diferentes tejidos vegetales ha sido reportada en diversas especies. Particularmente, los TFs DcMYB7 y DcMYB6 desempeñan un papel clave en el control de la pigmentación de las zanahorias moradas debida a la acumulación de antocianinas. En trabajos previos de nuestro grupo, se identificó dos transcriptos “antisentido” (asDcMYB6 y asDcMYB7) asociados a DcMYB6 y DcMYB7 y pudiendo potencialmente, participar en su regulación. En esta línea de trabajo proponemos probar de manera funcional, el mecanismo por el cual asDcMYB7 estaría ligado a la activación de la biosíntesis de antocianinas mediante el gen DcMYB7.